- 製品
- 製品
- CrossLab サポート商品
- サポート商品
- テクニカルサポート
- HOME
- 分野別ソリューション
- ゲノミクス
- SurePrint オリゴヌクレオチドライブラリ(オリゴプール)

※製品はすべて試験研究用です。診断目的にご利用いただくことはできません。
高品質で長いオリゴヌクレオチドライブラリの作製は、カスタムオリゴヌクレオチドの複雑なプールの合成から始まります。アジレントは、インクジェット技術に基づく独自のアレイベースの合成法 (SurePrint 技術)により、個々の塩基をスライド上の定義された位置に合成します。オリゴヌクレオチドはスライドから切り離され、プール後、凍結乾燥され、1本のチューブでお客様もとに届きます。オリゴライブラリを再懸濁し増幅することで、さまざまな下流のアプリケーションで使用することができます。

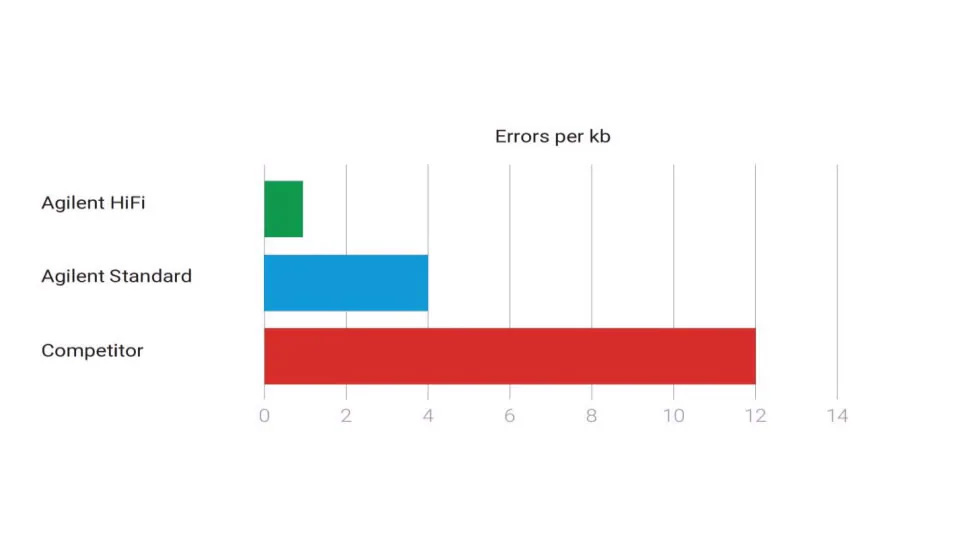
オリゴライブラリを選択する際に考慮すべき重要な要素は、FidelityとRepresentationです。アジレントのライブラリは、SurePrintプラットフォームで合成されます。Representationとは、プール内の各オリゴ配列の相対的な存在量のことです。Agilent SurePrint ライブラリは、Standardでは0.4%、HiFiでは0.1%とエラー率が低く、高いRepresentationを持っているため、より明確な結果の取得とスクリーニング時間の短縮につながります。
タンパク質、ゲノム、抗体工学、MPRA、遺伝子アセンブリ、その他多くのアプリケーションには、長鎖オリゴのプールDNAライブラリが不可欠です。アジレントは、長いオリゴの合成において技術的なスペシャリストでした。従来の合成法では、長さが50ヌクレオチド程度を超えると、多くの欠失が蓄積されるようになります。この時点で収量が低くなり、効率的にオリゴを合成・精製することができなくなります。SurePrint 技術を用いることで、230塩基までの長鎖オリゴの合成が可能となりました。